常態性檢定(Normality Test)
Shapiro-Wilk 檢定
- 說明:
R 已有內建 Shapiro-Wilk 檢定函式
shapiro.test()。 - 範例:
shapiro.test(<待檢定數據>) - 成果驗證:使用 R 與 SPSS 之結果相同,本結與 Kolmogorov-Smirnov 檢定一併展示。
Kolmogorov-Smirnov 檢定
- 說明:
R 已有內建 Kolmogorov-Smirnov 檢定函式
ks.test()。 - 範例:
ks.test(<待檢定數據>, "pnorm", mean = mean(<待檢定數據>), sd = sd(<待檢定數據>), alternative = c("two.sided")) # alternative 預設使用 "two.sided",可視需求選用 "less" 或 "greater"。 - 成果驗證:
使用 R 與 SPSS 之結果相同(p 值部分 R 預設功能不含 Lilliefors 校正,若必須使用可考慮使用 nortest 套件之
lillie.test()函式。另外,因本範例使用之樣本較少,故在 K-S 檢定時有警告信息):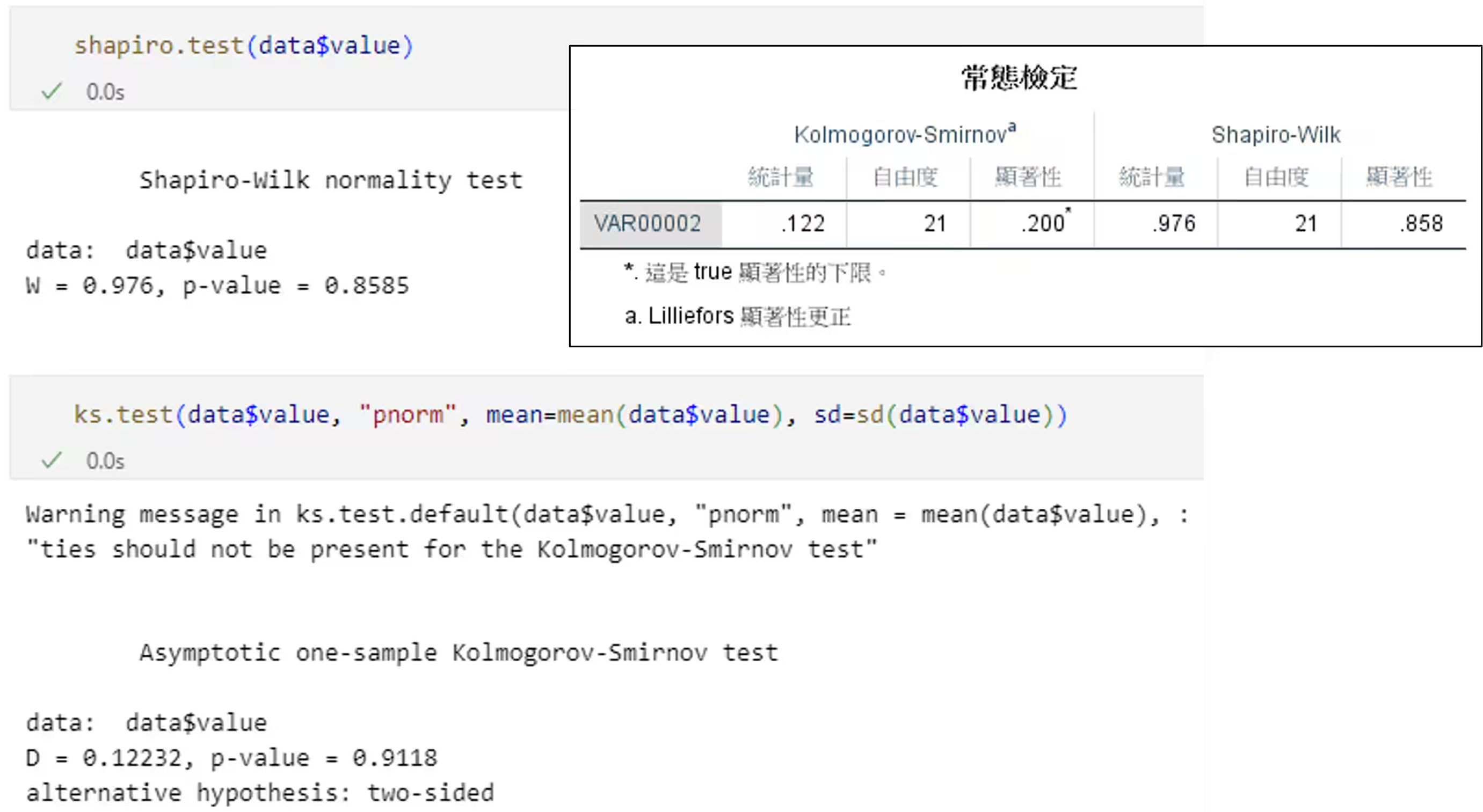
同質性檢定(Homogeneity Test)
Bartlett 檢定
- 說明:
R 已有內建 Bartlett 檢定函式
bartlett.test()。 - 範例:
bartlett.test(<待分析數據>~<待分析數據分組>, data = <數據來源>) # ex: bartlett.test(value~group, data = data)bartlett.test(<待分析數據>, <待分析數據分組>) # ex: bartlett.test(data$value, data$group)
Levene 檢定
- 說明:
R 通常通過 car 套件的
leveneTest()函式進行 Levene 檢定。 - 範例:
library(car) leveneTest(<待分析數據>~<待分析數據分組>, data = <數據來源>, center = mean) leveneTest(<待分析數據>~<待分析數據分組>, data = <數據來源>, center = median) # ex: leveneTest(value~group, data = data, center = median)library(car) leveneTest(y = <待分析數據>, group = <待分析數據分組>, center = mean) leveneTest(y = <待分析數據>, group = <待分析數據分組>, center = median) # ex: leveneTest(y = data$value, group = data$group, center = median) - 參考資料:
- 成果驗證:
使用 R 與 SPSS 之結果相同:
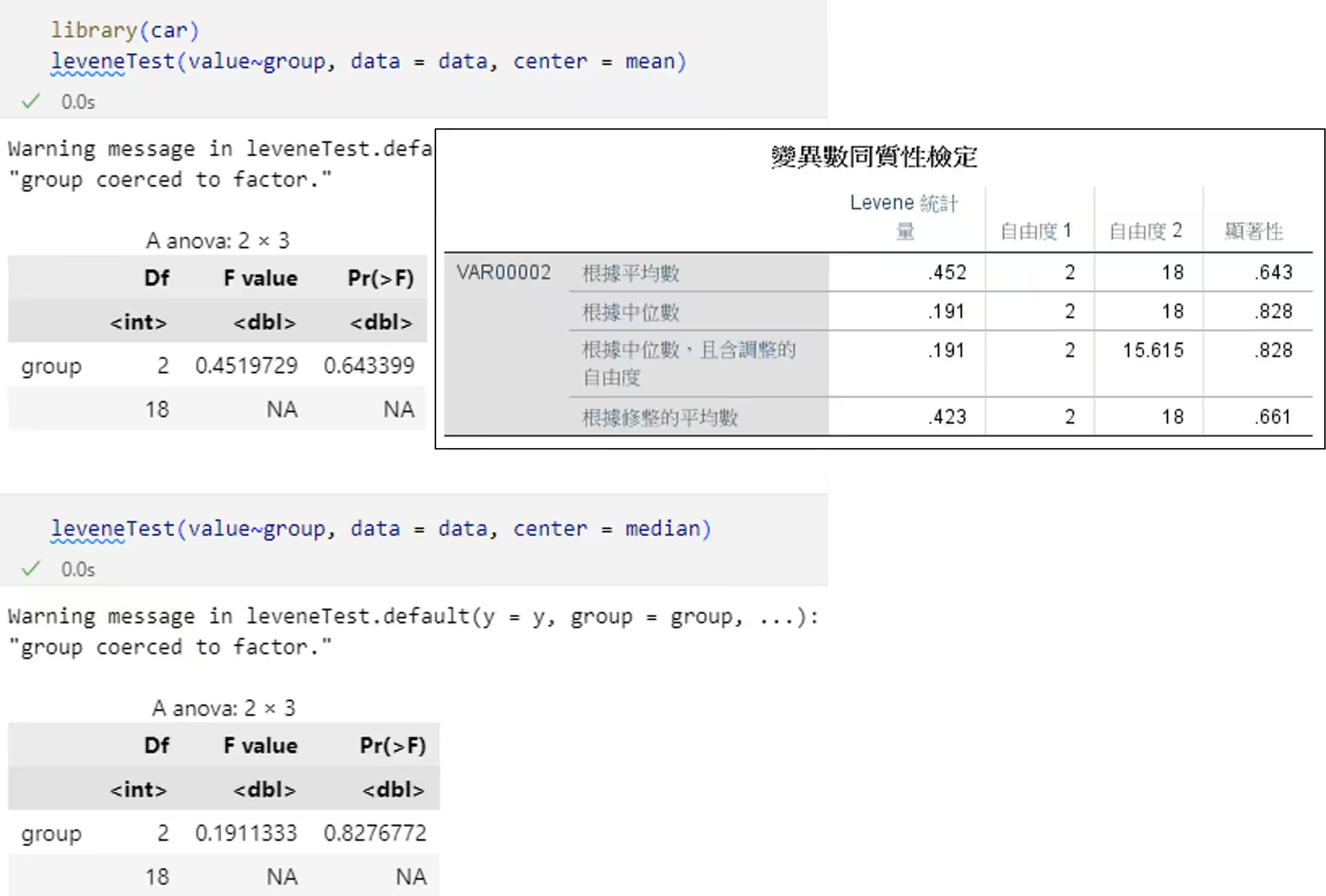
變異數分析
注意:進行變異數分析之前,務必先進行常態性檢定(Normality Test)以及變異數同質性檢定(Homogeneity Test for Variance),以確保符合統計分析方法之假設條件。
單因子變異數分析及其事後檢定
- 說明:
R 已有內建變異數分析函式
aov(),而事後檢定部分需透過其他套件實現,本範例使用 DescTools 套件進行單因子變異數分析之事後檢定。 - 範例:
library("DescTools") # 導入事後分析使用之 DescTools 函式庫。 data <- read.csv("<待分析檔案位址>", header = TRUE) # 載入待分析檔案。 # 透過單因子方差分析了解檔案中<待分析目標值>是否與<待分析組別>有關聯。 AnovaTest <- aov(<待分析目標值>~<待分析組別>, data = data) # ex: AnovaTest <- aov(value~group, data = data) summary(AnovaTest) # 檢視分析結果。 # 使用 DescTools/PostHocTest() 進行事後檢定,使用 Scheffé 法舉例。 # 方法可選 "hsd"、"bonf"、"lsd"、"scheffe"、"newmankeuls"。 PostHocTest(AnovaTest, method = "scheffe") - 參考資料:
- 成果驗證:
使用 R 與 SPSS 之結果相同(以 Scheffé 事後檢定為例):
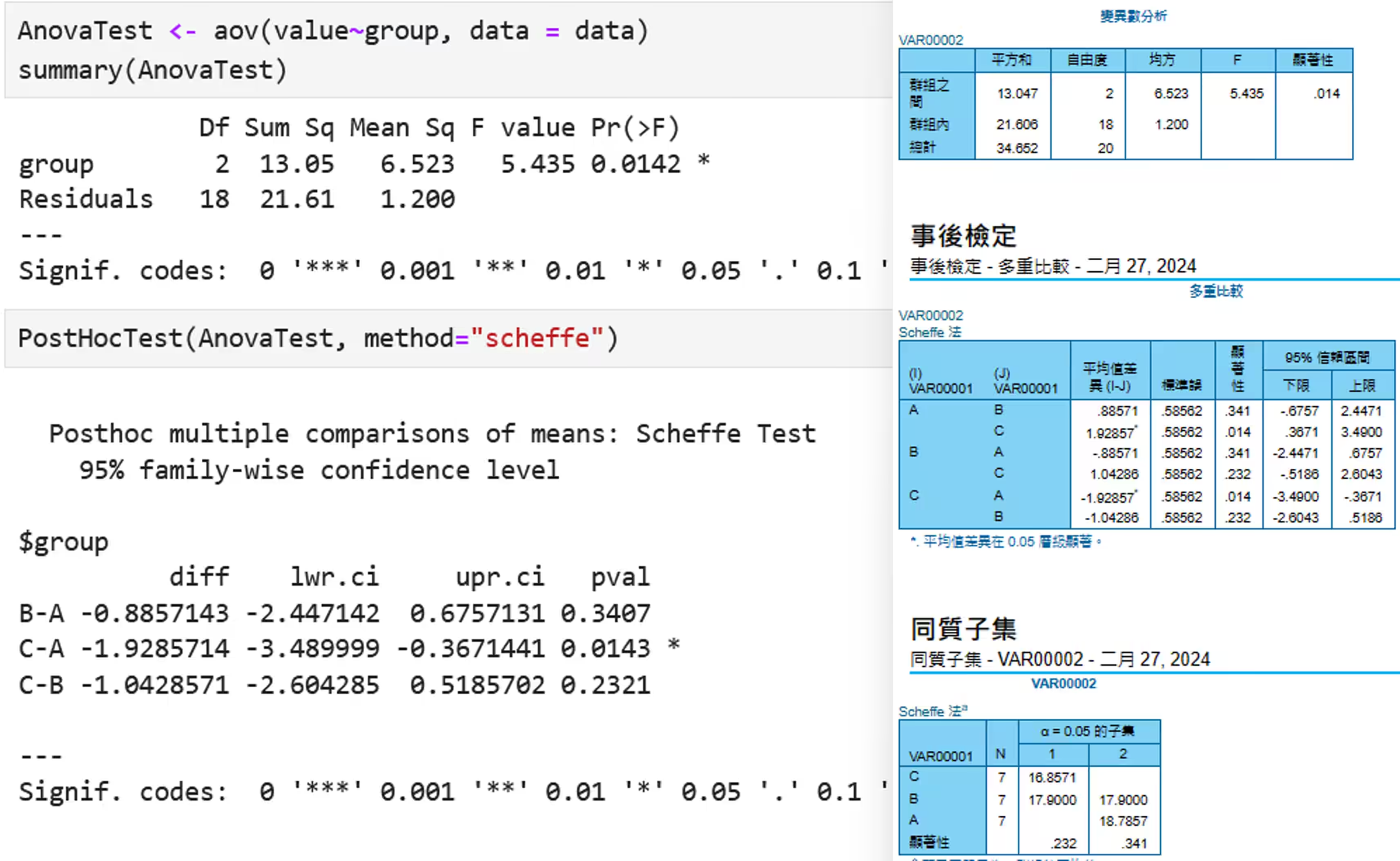
雙因子變異數分析及其事後檢定
29/02/2024 18:23
- 說明:
R 已有內建變異數分析函式
aov(),操作與單因子變異數同理。惟須特別注意,在進行變異數平方和計算時,有類型 I 至類型 IV 的差異,R 內建的aov()預設使用類型 I;SPSS 預設使用類型 III。若要在 R 中進行更細部的變異數分析可以考慮使用 car 套件的Anova()函式。然而,使用前建議慎重考慮是否真正知道何時應使用它,因為在 R 中相對沒有容易實現的方法。 - 範例:
- R/aov():
# 預設:類別 I library("DescTools") # 導入事後分析使用之 DescTools 函式庫。 data <- read.csv("<待分析檔案位址>", header = TRUE) # 載入待分析檔案。 AnovaTest <- aov(<待分析目標值>~<待分析組別 1> * <待分析組別 2>, data = data) # ex: AnovaTest <- aov(value~group1 * group2, data = data) summary(AnovaTest) # 檢視分析結果。 # 使用 DescTools/PostHocTest() 進行事後檢定,使用 Scheffé 法舉例。 PostHocTest(AnovaTest, method = "scheffe") # 類別 II data <- read.csv("<待分析檔案位址>", header = TRUE) # 載入待分析檔案。 aov(<待分析目標值>~<待分析組別 2> * <待分析組別 1>, data = data) aov(<待分析目標值>~<待分析組別 1> * <待分析組別 2>, data = data) # 此時,類別 II 的變異數平方和計算結果: # <待分析組別 1> 的結果為上式之結果; # <待分析組別 2> 的結果為下式之結果。 # 類別 III options(contrasts = c("contr.sum", "contr.poly")) data <- read.csv("<待分析檔案位址>", header = TRUE) # 載入待分析檔案。 AnovaTest <- aov(<待分析目標值>~<待分析組別 1> * <待分析組別 2>, data = data) # ex: AnovaTest <- aov(value~group1 * group2, data = data) drop1(AnovaTest, ~., test = "F") - car/Anova()(變異數計算使用類型 II 或 III):
library("car") # 類別 II Anova(lm(<待分析目標值>~<待分析組別 1> * <待分析組別 2>, data = data), type = 2) # ex: Anova(lm(分數~性別 * 身分別, data = data), type = 2) # 類別 III Anova(lm(<待分析目標值>~<待分析組別 1> * <待分析組別 2>, data = data, contrasts = list(<待分析組別 1> = contr.sum, <待分析組別 2> = contr.sum)), type = 3) # ex: Anova(lm(分數~性別 * 身分別, data = data, # contrasts = list(性別 = contr.sum, 身分別 = contr.sum)), type = 3)
- R/aov():
- 參考資料:
- 成果驗證:
- 使用 R/aov() 與 SPSS 所獲變異數分析結果相同,惟事後檢定結果稍有差異,我目前尚未深入了解其背後成因:
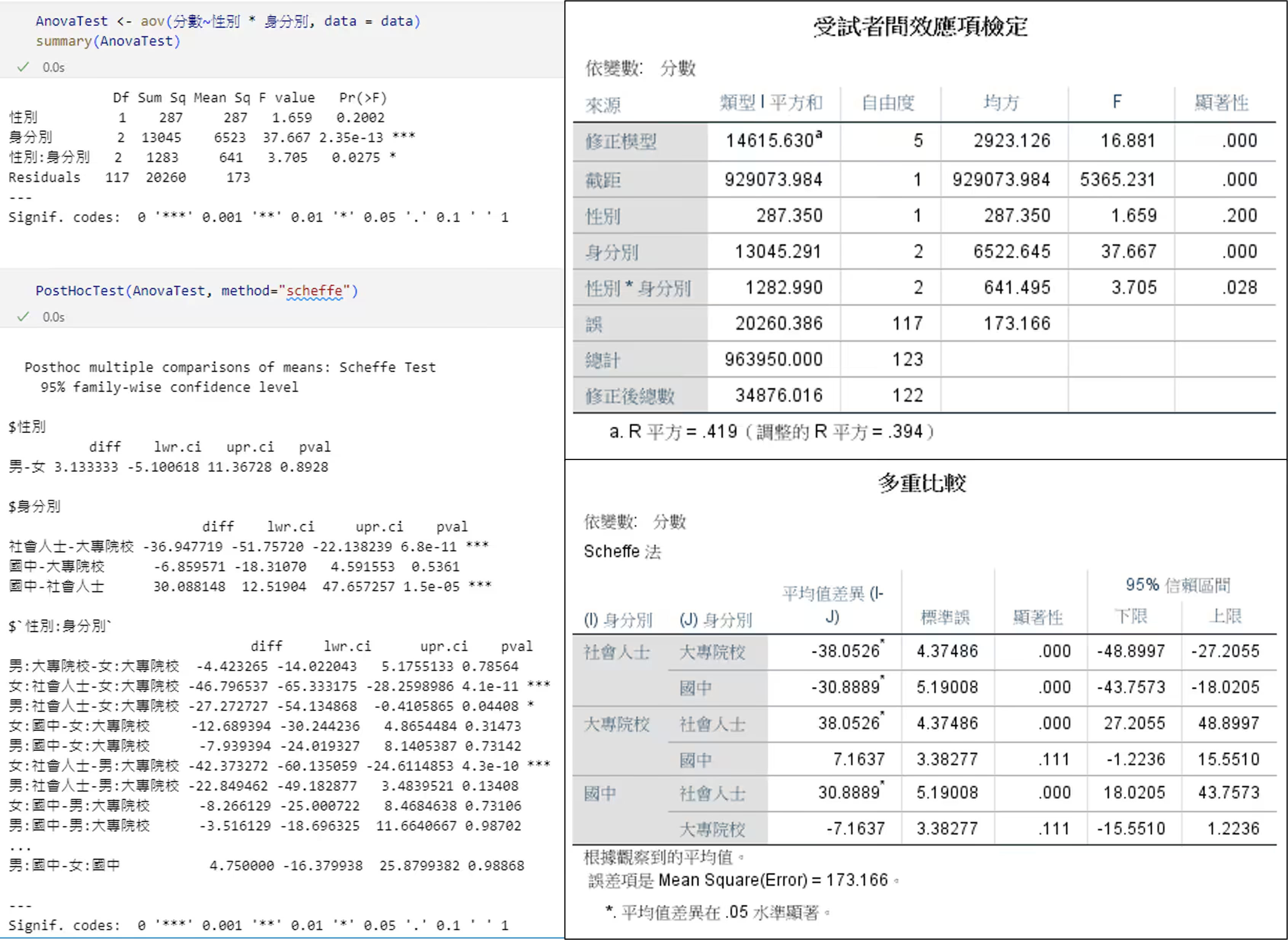
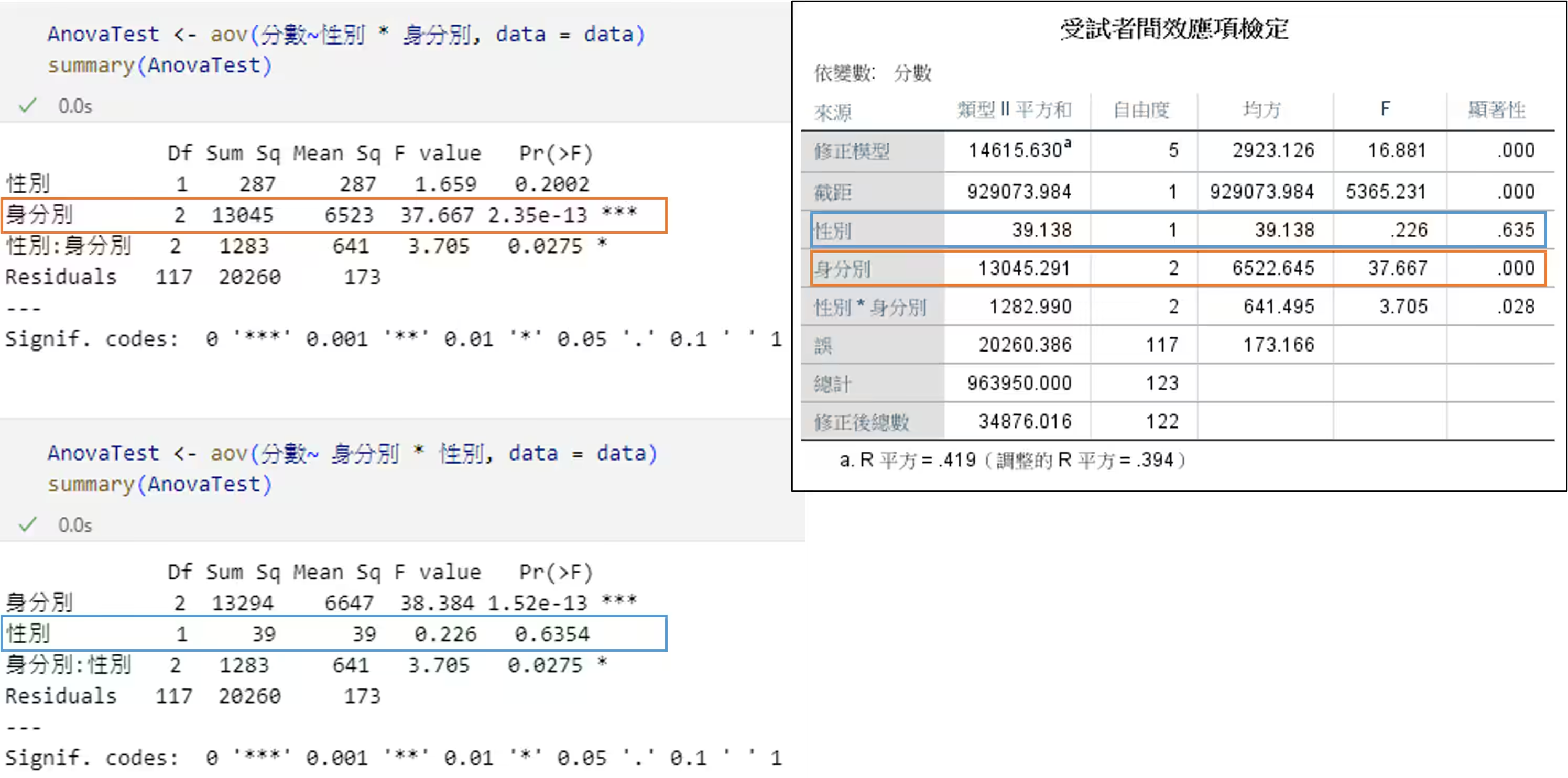
- 使用 car/Anova() 對於類別 II 與 類別 III 的變異數分析結果與 SPSS 所獲結果相同:
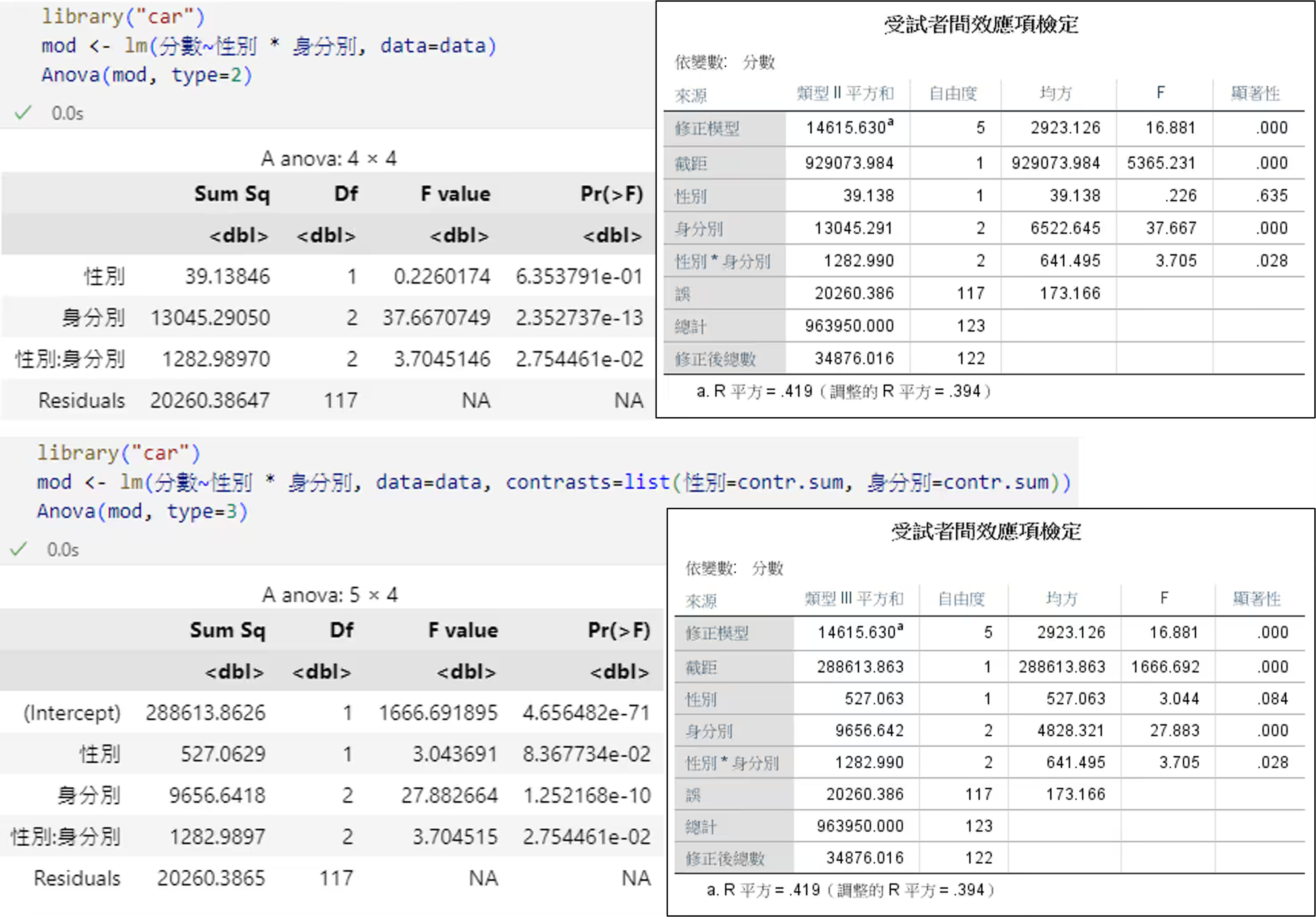
- 使用 R/aov() 與 SPSS 所獲變異數分析結果相同,惟事後檢定結果稍有差異,我目前尚未深入了解其背後成因:
Cronbach’s Alpha
23/03/2024 20:26
- 說明:
R 可透過 DescTools 套件之
CronbachAlpha()函式或是原生方法進行 Cronbach’s Alpha 之計算。 - 範例:
- DescTools/CronbachAlpha():
library("DescTools") # 導入 Cronbach’s Alpha 使用之 DescTools 函式庫。 data <- read.csv("<待分析檔案位址>", header = TRUE) # 載入待分析檔案。 # 透過 Cronbach’s Alpha 分析了解待分析目標項目數據之一致性。 CronbachAlpha(data[<待分析目標項目>], conf.level = <信賴水準 或 NA>) # ex: CronbachAlpha(data[c('q2_1', 'q2_2', 'q2_3'), conf.level = NA]) # ---補充: If item deleted--- CronbachAlphaIfItemDeleted <- function(data) { alpha <- CronbachAlpha(data) cat("Cronbach's Alpha for all items:", alpha, "\n") k <- ncol(data) alphas <- numeric(k) for (i in 1:k) { subset_data <- data[, -i] alphas[i] <- CronbachAlpha(subset_data)} names(alphas) <- colnames(data) cat("Cronbach's Alpha if item deleted:\n") print(alphas) } # 透過 Cronbach’s Alpha 分析了解待分析目標項目數據之一致性,以及若去除該項目後是否會變化。 CronbachAlphaIfItemDeleted(data[<待分析目標項目>]) # ex: CronbachAlphaIfItemDeleted(data[c('q2_1', 'q2_2', 'q2_3')]) - 原生 R 實現基本 CronbachAlpha() 計算,可應用於前一範例中之
CronbachAlphaIfItemDeleted():CronbachAlpha <- function(data) { k <- ncol(data) variances <- apply(data, 2, var) total_variance <- var(rowSums(data)) alpha <- (k / (k - 1)) * (1 - sum(variances) / total_variance) return(alpha) }
- DescTools/CronbachAlpha():
- 參考資料: